检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2016年2月23日,《细胞研究》(Cell Research)杂志在线发表了beat365官方网站生物动态光学成像中心汤富酬研究组、黄岩谊研究组和首都医科大学附属北京世纪坛医院暨北京大学第九临床医学院肝胆胰外科彭吉润研究组合作的最新研究成果——单细胞三重组学测序揭示肝癌细胞基因组、表观组和转录组的异质性(Single-cell triple omics sequencing reveals genetic, epigenetic, and transcriptomic heterogeneity in hepatocellular carcinomas)。该项研究建立了一种全新的单细胞三重组学测序方法(scTrio-seq),在国际上首次从同一个单细胞中实现对三种组学高通量测序信息的同时获取,并从单细胞水平发现肝癌细胞在三种组学上存在密切相互关联的高度异质性。
生物体内的同一种组织甚至同一种细胞类型的各个细胞之间,也往往存在着很大的差异。而传统的基于混合大量细胞的组学研究不仅无法观察到细胞之间的差别,并且会导致一部分重要细胞的独特特征被掩盖。因此单细胞组学分析技术对系统、深入研究特异种类的细胞特征以及细胞之间的差异起着重要作用。近年来,科学家们已经分别在基因组、转录组和DNA甲基化组等水平实现了单细胞单一组学的高通量测序分析,并且研究了单个生殖细胞、早期胚胎细胞以及癌症细胞的组学特征。
单细胞的多组学研究一直是科学家们的梦想。最近,国际上若干研究组分别报道了单细胞双组学同时测序技术(单细胞基因组和转录组; 单细胞DNA甲基化组和转录组),然而,目前一直无人能突破单细胞三个组学甚至更多个组学平行测序的技术瓶颈。
scTrio-seq技术首次实现了单细胞三重组学的高通量测序分析,可同时检测哺乳动物同一个单细胞内的基因组、DNA甲基化组和转录组信息。
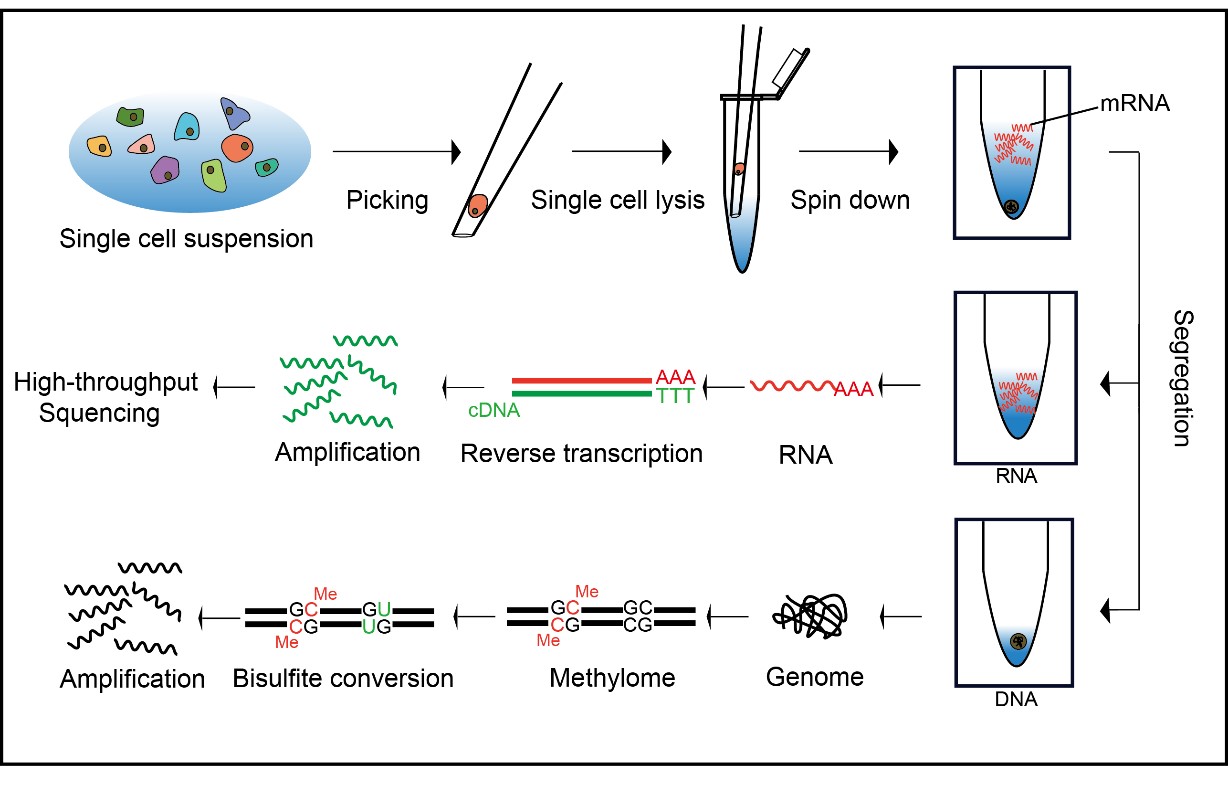
单细胞三重组学测序技术(scTrio-seq)
该项研究发现,在单个细胞内,基因区域DNA甲基化程度与基因表达有着很强的正相关性,且甲基化发生在不同的基因区域上会对基因的表达有不同的调控作用。接着,研究者们还发现基因组DNA的拷贝数与该区域上的基因表达亦有很强的正相关性,即DNA拷贝数较多的区域,其上的基因表达量也随之增高,反之亦然,从而在单细胞水平上证明了基因表达的剂量效应。然而DNA拷贝数的变化并不会影响其上DNA甲基化水平的变化。
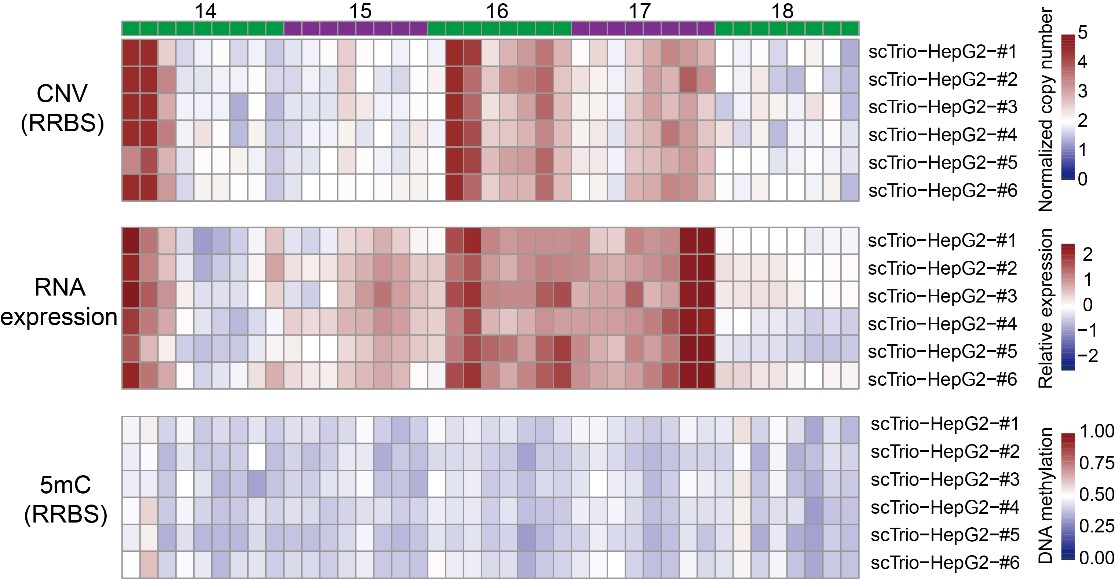
单细胞中三种组学之间的对应关系
我国是肝癌高发区,并且肝癌患者的预后较差,因此对肝癌的发生和转移的机制研究迫在眉睫。而癌症组织又是一个同时在基因组、转录组和表观组等层面上都存在高度异质性的细胞群体,因此更加需要采用单细胞三种组学同时分析技术来深入研究肿瘤发生过程中三种组学之间的调控机制。
该研究团队运用最新开发的scTrio-seq技术对一名肝细胞肝癌病人癌组织中的25个癌细胞进行了三种组学的同时分析。三种组学的数据都表明这25个肝癌细胞来自两个不同的细胞亚群。在此基础上,研究者们进一步分析了两个亚群在三种组学上的差异。研究发现,在基因组水平上,亚群I中的细胞含有较多拷贝数增加的染色体及染色体片段,而亚群II中含有较多拷贝数减少的染色体和染色体片段;在DNA甲基化水平上,亚群I比亚群II有更高的DNA甲基化水平,且产生的差异大多发生在CpG岛(CGI)区域;在转录组水平上,多个与炎症免疫应答相关的基因,以及与补体激活相关的基因的表达在亚群I中都有显著的降低,而与癌症发生和转移相关的重要基因ANO1和S100A11等的表达在两个亚群中亦有明显的差别。多种组学的数据显示,在被检测肝癌细胞中,数量上占比较小的亚群I细胞拥有更多的DNA拷贝数变异以及更高的DNA甲基化水平,可能更易逃脱患者免疫应答系统的识别,因而可能更加容易发生转移。
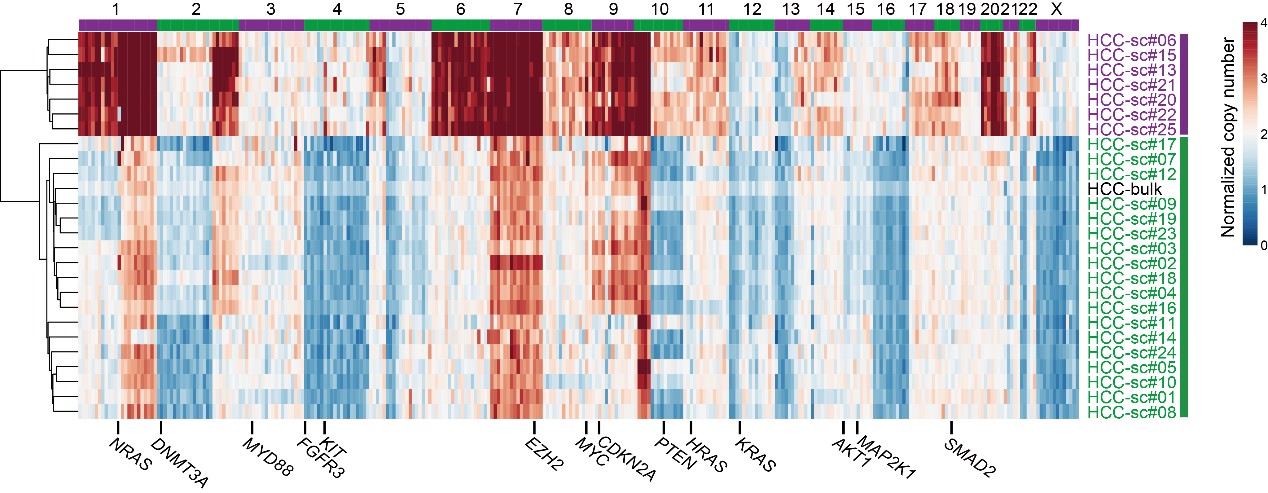
癌症中两个细胞亚群的DNA拷贝数差异
beat365官方网站生物动态光学成像中心的汤富酬研究员、黄岩谊研究员和北京世纪坛医院肝胆胰外科的彭吉润教授是该论文的共同通讯作者。beat365官方网站的博士研究生侯宇和北京大学医学部的硕士研究生郭化虎是这篇论文的并列第一作者。汤富酬研究员和黄岩谊研究员同时是北大清华生命科学联合中心的研究员。该项研究得到了国家自然科学基金和科技部的支持。
论文信息:
题目:Single-cell triple omics sequencing reveals genetic, epigenetic, and transcriptomic heterogeneity in hepatocellular carcinomas
杂志名称:Cell Research
链接:http://www.nature.com/cr/journal/vaop/ncurrent/full/cr201623a.html
DOI:10.1038/cr.2016.23