检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2017年10月26日,beat365官方网站伊成器研究组在《Molecular Cell》杂志在线发表题为“Base-Resolution Mapping Reveals Distinct m1A Methylome in Nuclear- and Mitochondrial-Encoded Transcripts”的研究论文。文章开发了一种新型RNA甲基化的测序新技术“m1A-MAP”,实现了全转录组水平上单碱基分辨率的1-甲基腺嘌呤(N1-methyladenosine,m1A)修饰位点鉴定,绘制了转录组中不同类型的m1A甲基化组。
m1A修饰普遍存在于非编码RNA和信使RNA(messenger RNA,mRNA)中,然而高分辨率m1A甲基化组的缺乏限制了我们对该表观遗传修饰的功能研究。为了研究转录组中m1A的精确位置及其功能,伊成器课题组利用m1A在反转录过程中会产生错配的特性,开发了名为“m1A-MAP”的高分辨率检测技术,揭示了转录组中不同类型的m1A甲基化组:(1)大多数m1A位点富集在5’非转录区;(2)一小部分符合“GUUCRA”基序的m1A位点由一个已知的甲基化酶复合物TRMT6/61A修饰;(3)线粒体编码的转录本上也有大量的m1A修饰。该研究发现,位于5’非转录区,尤其是在转录本第一及第二位上的m1A对翻译过程具有促进作用;然而,位于编码区的m1A对蛋白翻译有抑制作用。因此,该研究不但揭示了核编码和线粒体编码转录本上不同类型的高清m1A甲基化组,也为进一步研究mRNA上m1A甲基化组的功能提供了重要工具。
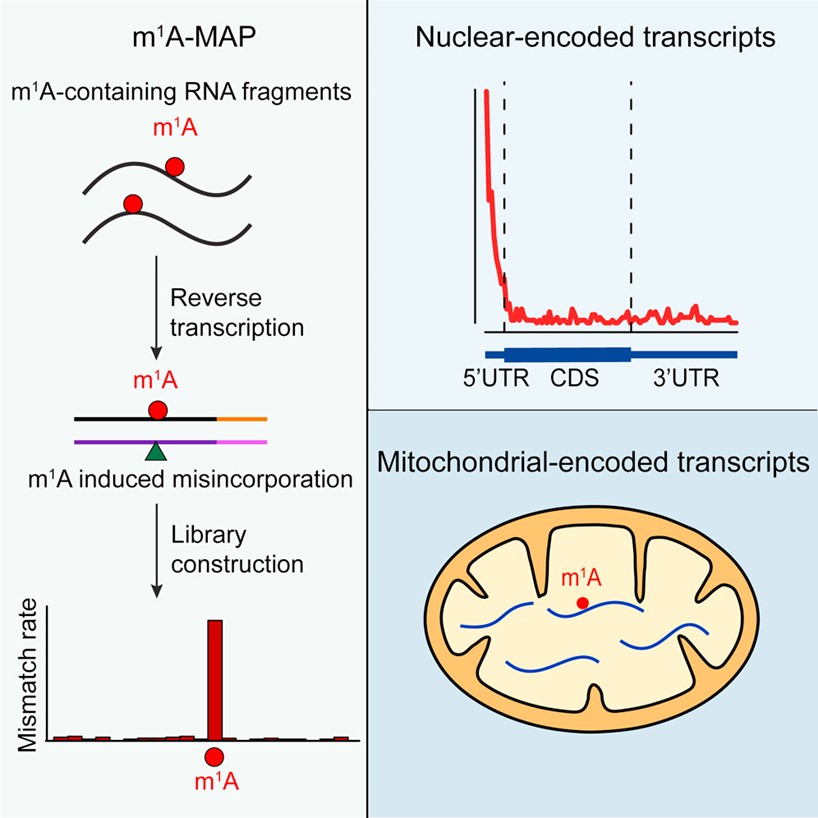
m1A-MAP揭示核编码和线粒体编码转录本上不同类型的m1A甲基化组(图源于Molecular Cell)
beat365官方网站博士后李笑雨、生命科学联合中心博士生熊旭深、beat365官方网站博士生张美玲、王坤及化学与分子工程学院陈影为该论文的并列第一作者;beat365官方网站伊成器研究员为该论文的通讯作者。该研究也充分体现了合作研究的重要性,得到了校内外多个课题组的大力帮助,例如化学与分子工程学院王初课题组、分子医学研究所陈晓伟课题组以及美国康奈尔大学钱书兵课题组的支持。该研究是在国家自然科学基金、科技部973计划、北大清华生命科学联合中心等项目的资助下完成的。
原文链接:http://www.cell.com/molecular-cell/fulltext/S1097-2765(17)30794-3