检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2018年3月6日,beat365官方网站、北大-清华生命科学联合中心陆剑课题组在国际知名学术期刊《RNA》上以长文形式在线发表题为“MicroRNA Duplication Accelerates the Recruitment of New Targets During Vertebrate Evolution” (doi:10.1261/rna.062752.117)的研究论文。该论文报道了脊椎动物中miRNA复制之后发生的功能分化及miRNA复制在进化过程中对新靶位点招募的促进作用。
miRNA是一类生物体内源表达的长约22碱基的非编码小RNA,通过种子序列互补配对的方式识别靶基因mRNA并调控其表达。由于这种较短的配对方式,一种miRNA通常有上百个靶基因,一个靶基因也可以同时被多个miRNA调控。这种“多对多”的作用方式决定了miRNA:mRNA相互作用调控网络的复杂性。那么miRNA是如何起源和演化的呢?早在2008年,陆剑研究员便开始从演化生物学角度研究动物中非编码小RNA的调控机理和演化模式。他提出的“出生-死亡”模型,揭示了绝大部分新miRNA在演化中快速产生和快速死亡 (Lu et al. 2008a),并发现正向选择是果蝇中新miRNA产生的驱动力(Lu et al. 2008b),之后在群体基因组学水平上系统地研究了miRNA调控系统变异和人类转录组多态性的关系 (Lu & Clark, 2012)。
那么,哪些特征可以使新产生的miRNA在演化过程中更容易被保留下来呢?近年来陆剑课题组对这一问题进行了研究。基因组中很多miRNA是成簇存在的,形成多顺反子结构被共同转录。陆剑课题组发现同簇不同家族的miRNA在功能上有趋同进化的现象,并提出了“功能共适应模型”来说明成簇存在的miRNA在演化过程中更容易积累适应性突变并被自然选择所保留下来 (Wang et al. 2016)。在刚刚发表的这项研究中,陆剑课题组通过系统性地研究人类不同组织中小RNA的表达谱,并结合靶基因的预测以及已发表的实验数据,发现基因复制产生的旁系同源miRNA在基因表达和靶基因偏好上存在广泛差异,具有相同种子序列的旁系同源miRNA(Duplicated miRNAs)比单拷贝的miRNA拥有显著更多的靶基因。通过比较基因组学分析,他们认为miRNA复制为新产生的靶位点提供了更为广泛的共进化环境,从而使那些靶位点更容易产生功能并在进化过程中保留下来。
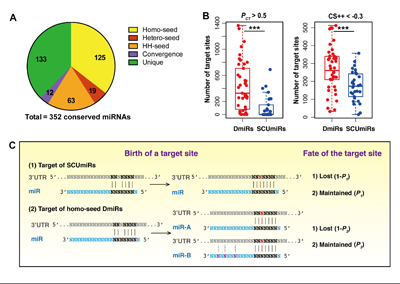
图. microRNA复制之后的功能分化及与靶位点的协同进化
A.人类基因组中各种不同类型保守miRNA的数目。B.旁系同源多拷贝miRNA比单拷贝的miRNA 拥有显著更多的靶基因。C.旁系同源多拷贝miRNA与单拷贝miRNA的靶基因进化过程差异比较示意图。
通过在四种哺乳动物(人,恒河猴,非洲绿猴,大鼠)的肾源细胞系中过表达let-7a和let-7b,结合高通量测序和转录组深度分析,进一步验证了miRNA复制之后不同拷贝对靶基因有不同的偏好性,并发现let-7靶位点的获得和丢失与不同物种间基因表达演化相关。
陆剑研究员为该论文的通讯作者。生科院博士后罗俊杰(现为中国农业大学北京市食品营养与人类健康高精尖创新中心岗位科学家)、生命科学联合中心博士生王奕蓉、袁健是论文的共同第一作者,生科院本科生赵志磊(现就读于普林斯顿大学)参与了该研究。该工作得到了国家科技部、国家自然科学基金委以及北大-清华生命科学联合中心的资助。
原文链接:
http://rnajournal.cshlp.org/content/early/2018/03/06/rna.062752.117.abstract
https://academic.oup.com/mbe/article/33/9/2232/2578711
参考文献
Lu J, Shen Y, Wu Q, Kumar S, He B, Shi S, Carthew RW, Wang SM, Wu CI. 2008a. The birth and death of microRNA genes in Drosophila. Nat Genet 40:351-355.
Lu J, Fu Y, Kumar S, Shen Y, Zeng K, Xu A, Carthew R, Wu CI. 2008b. Adaptive evolution of newly emerged micro-RNA genes in Drosophila. Mol Biol Evol 25:929-938.
Lu J, Clark AG. 2012. Impact of microRNA regulation on variation in human gene expression. Genome Res 22:1243-1254.
Wang Y, Luo J, Zhang H, Lu J. 2016. microRNAs in the Same Clusters Evolve to Coordinately Regulate Functionally Related Genes. Mol Biol Evol 33:2232-2247.