检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
红细胞是人体中数目最多的细胞,也是体内最活跃的自我更新体系,健康人每日需更新约二十亿个红细胞。红细胞发育过程的缺陷会导致许多贫血或再生不良的疾病发生,例如先天性纯红再障(DBA)等[1-3],因此理解红细胞发育各阶段的生物学事件及其机制是极为重要的。红细胞发育后期经历一系列细胞生物学的显著变化,包括染色质压缩、退出细胞周期等,哺乳动物的红细胞最终还要经历“脱去细胞核”的关键步骤,以产生新生红细胞。红细胞终末发育的染色质逐步压缩过程中仍然需要保持血红蛋白基因等红细胞功能相关基因的表达,此过程中的细胞如何在染色质高度压缩的状态下维持基因表达、染色质压缩过程是否有一定的程序性规律与调控机制一直是悬而未决的问题[4]。
2023年3月13日beat365官方网站、北大-清华生命科学联合中心、北京大学血液病研究所李湘盈课题组在Nature Structural & Molecular Biology 发表题为“Heterochromatin rewiring and domain disruption-mediated chromatin compaction during erythropoiesis”的研究论文,该研究首次报道了人类红细胞终末分化过程中,异染色质的大规模重构可能是染色质压缩的主要贡献力量,且染色质拓扑相关结构域(topologically associating domain, TAD)发生大规模瓦解,转录因子GATA1参与到未瓦解TAD的维持过程中。这将为理解和促进红细胞相关疾病研究提供理论基础。

首先,作者在人类原代红系培养中分离出高纯度早期成红细胞和晚期成红细胞,而后综合利用Hi-C、HiChIP及多种组学技术,首次解析了人红细胞终末发育不同时期的染色质压缩过程。异染色质区域和活跃染色质区域展示出不同的压缩特征: 伴随着全局的染色质压缩,异染色质区域形成超长的区室间的相互作用; TAD 发生较大规模的瓦解,这种瓦解不仅与 TAD 边界 CTCF 及 SMC3 结合强度减弱相关,而H3K27ac 及GATA1结合的活跃染色质区域与终末发育阶段TAD的完整性具有相关性。本研究中发现并功能性验证红细胞重要转录因子GATA1参与到红系终末分化过程中的染色质结构域维持以及终末时期转录作用。此外有意思的是,脱出的细胞核仍维持较完整的染色质结构。
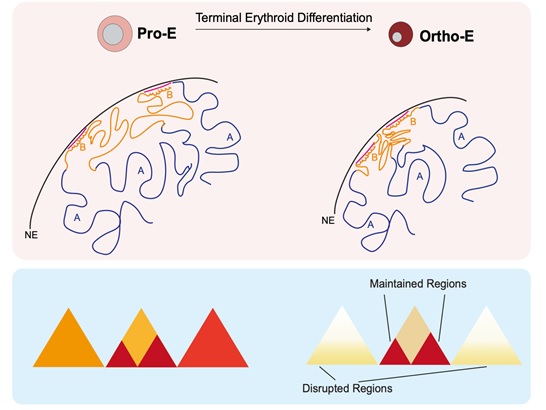
图1 红细胞终末分化染色质结构改变示意图
综上所述,本研究利用多种染色质结构解析技术,解析了人类红细胞终末分化染色质压缩的一般规律及异染色质/活跃染色质相互作用在染色质压缩中的主要角色; 揭示了GATA1通过保持红系终末分化中染色质结构域完整调控红细胞分化过程的潜在新功能。本研究为更好的理解染色质高级结构改变特别是染色质压缩提供了理论基础,同时也为红细胞终末发育相关疾病的研究提供了新的思路。
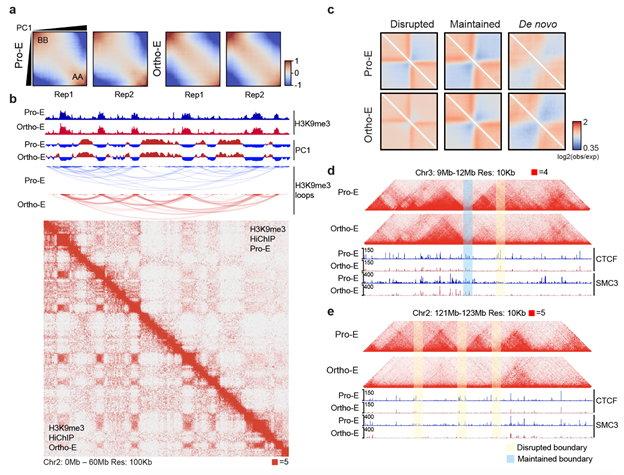
图2 红细胞终末分化过程中长距离异染色质互作形成(a-b),拓扑相关结构域-TAD瓦解(c-e)
beat365官方网站、北大-清华生命科学联合中心、北京大学血液病研究所李湘盈研究员为本论文通讯作者。beat365官方网站博士后(原清华大学博士研究生)李栋及beat365官方网站博士研究生吴帆为该论文的第一作者,beat365官方网站博士研究生周硕参与了部分工作。国家血液系统疾病临床医学研究中心、北京大学人民医院、北京大学血液病研究所黄晓军教授合作参与完成了这一工作,并给予了大力支持。
参考文献:
1. Dzierzak, E. and S. Philipsen, Erythropoiesis: development and differentiation. Cold Spring Harb Perspect Med, 2013. 3(4): p. a011601.
2. Palis, J., Primitive and definitive erythropoiesis in mammals. Front Physiol, 2014. 5: p. 3.
3. Sankaran, V.G. and M.J. Weiss, Anemia: progress in molecular mechanisms and therapies. Nat Med, 2015. 21(3): p. 221-30.
4. Keerthivasan, G., A. Wickrema, and J.D. Crispino, Erythroblast enucleation. Stem Cells Int, 2011. 2011: p. 139851.
原文链接:https://www.nature.com/articles/s41594-023-00939-3