检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2019年1月25日,魏文胜课题组在Genome Biology杂志在线发表了题为“Guide RNAs with embedded barcodes boost CRISPR-pooled screens”的研究论文。
CRISPR/Cas9系统作为强大的基因组编辑工具被广泛运用于基因的功能性筛选研究。魏文胜课题组在2014至 2018年先后实现了针对蛋白质编码基因和长链非编码RNA的高通量功能性筛选(Zhou et al. Nature 2014; Zhu et al. Nature Biotechnology 2016; Liu et al. Nature Biotechnology 2018)。为保证筛选结果的准确性,需要避免混合型文库中的“搭车效应”。目前通行的方法要求在慢病毒侵染构建文库时保证较低的感染复数(MOI);同时为获得高度可重复性的结果,需要多组实验重复。这些要求使常规筛选工作量巨大;同时当细胞来源受限时(如原代细胞),难以实现大规模的功能性筛选研究。
为解决以上难题,魏文胜课题组建立了允许高感染复数构建CRISPR文库的新方法。重新设计的sgRNA被赋予多条分子条码,被命名为iBAR (internal barcode)(图1)。这些内置分子条码能够多次、独立追踪sgRNA富集程度,结合重新适配的MAGeCKiBAR的生物信息学分析方法,成功避免了在高感染复数条件下的“搭车效应”,并提高了数据可靠性。与常规筛选比较,iBAR方法提高了功能性筛选的敏感性,显著降低由于高感染复数建库筛选中产生的高假阳性和假阴性。该研究成果首次实现了在高MOI条件下的高通量功能性筛选,显著降低了工作量,提高了筛选准确性,为在原代细胞或动物体内筛选提供了重要工具。
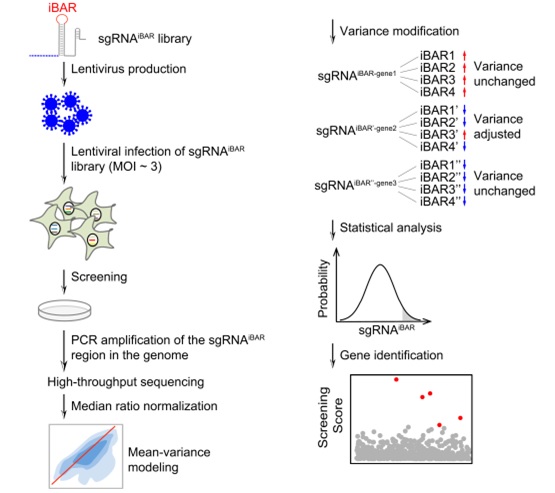
图1:CRISPR iBAR文库筛选分析流程示意图
魏文胜课题组的国家博新计划博士后朱诗优、博士生曹中正(CLS 15级)、刘志恒(CLS 15级)以及何苑(生命学院16级)为该论文共同第一作者。该研究项目得到了国家自然科学基金重点项目、北京未来基因诊断高精尖创新中心、北大-清华生命科学联合中心以及传染病防治国家科技重大专项的基金支持。
文章链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-019-1628-0